Institut für
FG Forstgenetik
Genomforschung
Im Arbeitsbereich Genomforschung verwenden wir genomische, molekularbiologische und bioinformatische Methoden, um die genetische Grundlage von anpassungsrelevanter Variation zu identifizieren und zu charakterisieren. Wir sequenzieren sowohl DNA als auch RNA von einer großen Anzahl von Individuen, um Sequenz- und Expressionsunterschiede zu detektieren. Mithilfe dieser umfangreichen Datensätze erfassen wir die natürliche genetische Variation und deren geografische Verteilung und bestimmen Assoziationen mit wichtigen Merkmalen und Umweltvariablen. Wir arbeiten außerdem daran, moderne Ansätze, wie z.B. Methoden der Genomeditierung, die Sequenzierung mit ultra-langen Reads oder die Pangenomik, in verschiedenen Waldbaumarten zu etablieren und anzuwenden. Zusammenfassend soll unsere Arbeit dazu beitragen, neue wissenschaftliche Erkenntnisse zu generieren und Entscheidungshilfen für die Politik bezüglich der Klimaanpassung der Wälder zu entwickeln.
Natürliche genetische Variation
Mutationen und die daraus resultierende natürliche genetische Variation sind die Grundlage der Evolution, der Domestizierung und der Züchtung. Natürliche Genvarianten bergen ein enormes für die Anpassung und den Erhalt von Waldökosystemen. Beispielsweise können natürliche Resistenzen zum Schutz von Baumarten beitragen, die von eingeschleppten Schädlingen oder Pathogenen bedroht sind, so wie die Esche vom Eschentriebsterben. Dafür benötigen wir allerdings ein genaues Verständnis der zugrundeliegenden Genetik. Die molekulargenetische Untersuchung von evolutionär wichtigen Merkmalen (wie z.B. Zweihäusigkeit) kann grundlegende biologische Prozesse aufklären und damit ebenfalls wertvolle Informationen für die forstliche Praxis zur Verfügung stellen. Schließlich kann die Charakterisierung der genomweiten adaptiven Variation einen wichtigen Beitrag für die Anpassung unserer Wälder im Klimawandel leisten.
Ausgewählte Projekte:
PangenomBuche
Evolution Monözie Diözie ("MonDiö")
Buchengenomik
Ansprechpartner: Niels A. Müller
Bioinformatik
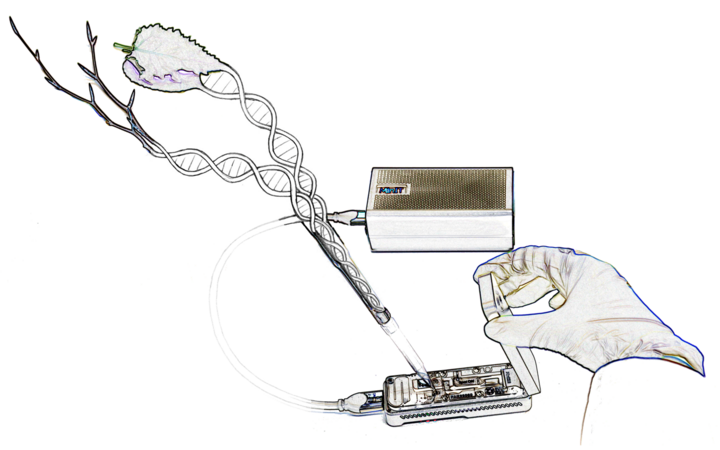
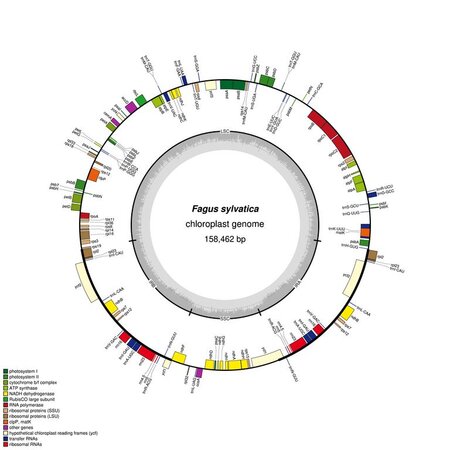
Moderne Hochdurchsatzsequenzierungen liefern eine außerordentlich große Menge an Genom- und Transkriptomdaten, die bioinformatisch ausgewertet werden müssen. Diese Auswertungen tragen dazu bei, verschiedene Fragestellungen innerhalb der funktionellen Genomforschung zu beantworten und diagnostische Marker zu entwickeln. Ein wichtiges Instrument hierbei ist die genomweite Detektion und der Vergleich genetischer Varianten, insbesondere von SNPs in phänotypisierten Individuen. Für die Entwicklung genetischer Marker zur Artdifferenzierung werden komplette Chloroplasten- und Mitochondriengenome verglichen, die für einige Baumarten neu assembliert und annotiert werden. Zur Unterstützung der Forschung im Institut werden wissenschaftliche Datenbanken und bioinformatische Tools entwickelt.
Ausgewählte Projekte:
TaxGen
Survivor-Oaks
Eichenabwehr
Holz-DNA-Barcoding
Datenbankentwicklung
Ansprechpartnerin: Birgit Kersten
FNR-Nachwuchsforschergruppe
Genetische Technologien
Die Methoden zur Genomeditierung, wie CRISPR/Cas, sind in Kulturpflanzen maßgeblich entwickelt worden. Für die Anwendung in unterschiedlichen Baumarten sind Weiterentwicklungen notwendig, die insbesondere zur Erforschung der Trockenstresstoleranz von Bäumen eingesetzt werden sollen.
Ausgewählte Projekte:
Weitere Informationen auf der Gruppenseite.
Ansprechpartner: Tobias Brügmann
Arbeitsbereichsleiter
Wissenschaftliche Mitarbeiter*innen
Doktorand*innen
Technisches Personal
Wissenschaftlich – „Peer-reviewed”
- Lazic D et al. (2024) Genomic variation of European beech reveals signals of local adaptation despite high levels of phenotypic plasticity. Nature Communications 15 (1), 8553. link
- Leite Montalvão AP et al. (2022) ARR17 controls dioecy in Populus by repressing B-class MADS-box gene expression. Philosophical Transactions of the Royal Society B 377 (1850), 20210217. link
- Renner SS, Müller NA (2021) Plant sex chromosomes defy evolutionary models of expanding recombination suppression and genetic degeneration. Nature Plants 7 (4), 392-402. link
- Müller NA et al. (2020) A single gene underlies the dynamic evolution of poplar sex determination. Nature Plants 6 (6), 630-637. link
Zielgruppenorientiert – Wissenschaftskommunikation
- Brügmann T, Zahn V, Fendel A, Fladung M (2025) Biotechnologische Innovationen an Rotbuchen und weiteren Baumarten. Thünen Rep 119:211-216. link
- Müller NA, Fladung M, Welling M (2022) Pappelsex: Zwei Geschlechter, ein Gen. Wissenschaft erleben. link
- Deecke K & Fladung M (2021) Melampsora-Resistenztests in Pappeln. AFZ-DerWald. link
- Brügmann T & Fladung M (2019) Genom-Editierung in Bäumen. AFZ-DerWald. link