
Institut für
FG Forstgenetik
Projekt
TreeHarm
Entwicklung von Diagnose- und Behandlungsverfahren zur Identifizierung von und Schutz einheimischer Bäume gegen geregelte und neue Schadorganismen
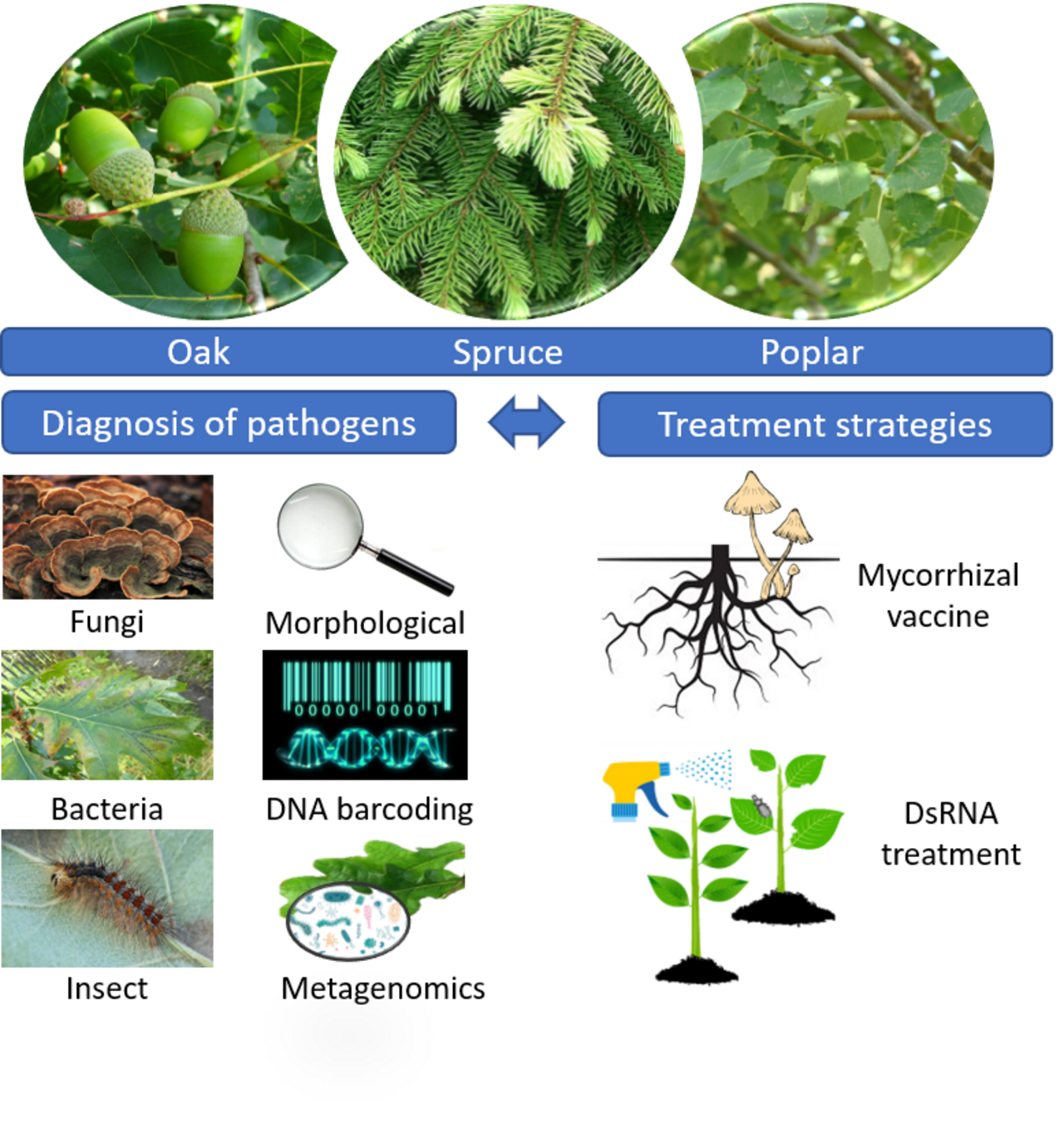
Das vom Bundesministerium für Ernährung und Landwirtschaft über die Fachagentur für Nachwachsende Rohstoffe (FNR) für drei Jahre geförderte Projekt „TreeHarm“ hat sich zum Ziel gesetzt, exemplarisch an der Eiche, der Fichte und der Pappel in Form einer „Hand-in-Hand“-Strategie zu prüfen, in wieweit eine phänotypische und/oder molekulare Identifizierung von Schaderregern (Insekten, Pilze, Bakterien) eine möglichst frühzeitige Initiierung von Behandlungsmaßnahmen ermöglicht.
Hintergrund und Zielsetzung
Der Klimawandel führt zu steigenden Temperaturen, trockenen und heißeren Sommern und verursacht Trockenstress für die Waldbäume. Der Trockenstress der Bäume gilt als Einladung für die Krankheitserreger. Überraschenderweise machen sich neue Pflanzenpathogene den Trockenstress zunutze und schädigen die Waldbäume (https://www.dw.com/de/deutschland-waldsterben/a-54330242). Das Schleswig-Holstein verfügt zwar über ~173,000 Hektar Waldfläche, ist aber dennoch das waldärmste Gebiet in der Bundesrepublik Deutschland (https://www.schleswig-holstein.de/DE/Fachinhalte/W/wald/wald_01_Allg_01_WaldSH.html). Es besteht daher ein dringender Bedarf, sowohl infektiöse als auch nützliche Lebensräume an einheimischen Waldbäumen des Landes Schleswig-Holstein zu identifizieren.
Vorgehensweise
Die Diagnose der Pilz-, Insekten- und Bakterienarten der drei Waldbaumarten wird mit DNA-Barcoding-, Metagenomik- und Morphotyping-Methoden durchgeführt. Die Genomregionen Cytochrom c Oxidase 1 Gen (“COX1”) (Insekten) und Internal Transcribed Spacer (ITS) Barcoding Regionen (https://ibol.org/) werden zur Identifizierung von Insekten- bzw. Pilzarten untersucht. Für die Identifizierung von Bakteriengemeinschaften wird hingegen das "INVIEW Metagenome Explore", eine Hochdurchsatz-Sequenzierungsmethode der nächsten Generation der Firma Eurofins Genomics, verwendet.
Für die Behandlung werden zwei alternative Strategien entwickelt und auf ihre Praxistauglichkeit getestet: die “Mykorrhiza-Impfung” und die RNA-Interferenz (RNAi). Das Verfahren “Mykorrhiza-Impfung” wird seit rund 100 Jahren erfolgreich in Baumschulen eingesetzt. In einem Feldversuch soll getestet werden, ob eine gezielte Anwendung von einheimischen Mykorrhizapilzen (Impfung) die individuelle Körperkonstitution der Bäume stärken kann.
Darüber hinaus wird die RNA-Interferenz (RNAi) basierend auf der Behandlung mit doppelsträngiger RNA (dsRNA) gegen Insekten- und Pilzpathogene getestet. Dabei wird doppelsträngige RNA (dsRNA) mit einer Länge von 18 bis 26 Nukleotiden auf mit Schädlingen (Pilzen, Insekten) befallene Pflanzenteile (Blätter, Triebe) mit Hochdruck aufgesprüht. Die dsRNA, die bei diesen Erregern zu Teilen der Sequenz lebenswichtiger Gene komplementär ist, kann die Expression dieser Gene durch Interferenz hochspezifisch hemmen oder ganz ausschalten. Die Vorteile der “RNA-Impfung” gegenüber herkömmlichen Pestiziden sind vielfältig: (i) Hohe Spezifität: die Abwehr des Erregers ist sehr spezifisch, (ii) keine Rückstände: RNA-Moleküle werden sehr schnell abgebaut, (iii) keine Resistenz: Es ist fast unmöglich, dass Schädlinge und Krankheitserreger gegen diese Art der Bekämpfung resistent werden.
Thünen-Ansprechperson

Thünen-Beteiligte
Geldgeber
-
Fachagentur Nachwachsende Rohstoffe e.V. (FNR)
(national, öffentlich)
Zeitraum
4.2021 - 3.2024
Weitere Projektdaten
Projektstatus:
abgeschlossen

