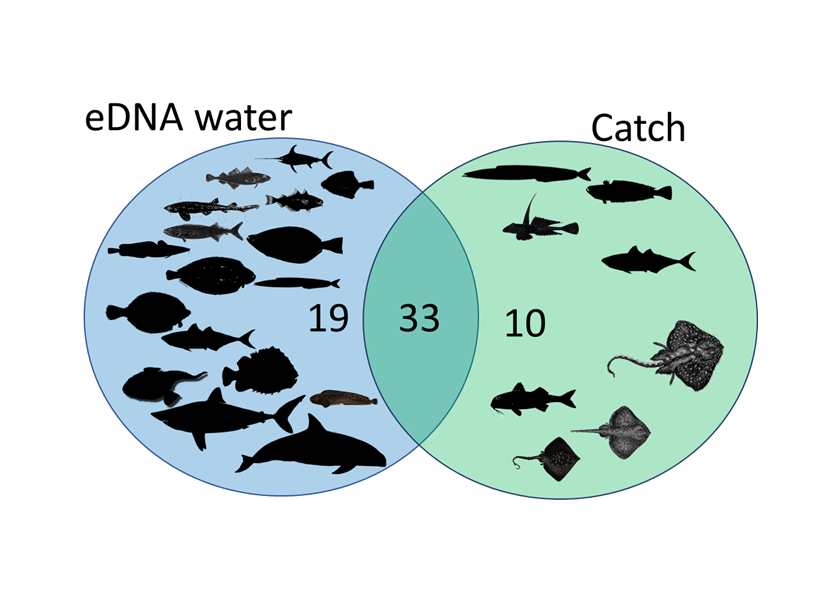
Eine vollständige Erfassung der Fischvielfalt in verschiedenen Lebensräumen des Meeres ist nach wie vor eine Herausforderung für die Wissenschaft. Ein Forscherteam der Thünen-Institute für Fischereiökologie und für Seefischerei um Yassine Kasmi, Reinhold Hanel und Ismael Núñez-Riboni testete dafür die Analyse von Umwelt-DNA (eDNA) in der Nordsee als neue und nicht invasive Monitoring-Methode. Die Untersuchung verglich klassische Grundschleppnetzfänge mit einer genetischen Analyse von Wasser- und Sedimentproben. Dabei zeigte sich: eDNA aus Wasserproben erkennt bis zu 81 % der Arten, die auch durch klassische Fangmethoden erfasst werden und identifizierte sogar zusätzliche, mit Schleppnetzen schwer erfassbare Arten wie Haie oder Schweinswale.
Die Studie ging noch einen Schritt weiter: Für die in der Nordsee häufige Kliesche (Limanda limanda) wurde versucht, aus der Anzahl der genetischen Spuren auf deren Biomasse an bestimmten Standorten zu schließen. Das Ergebnis ist vielversprechend – unter Einbezug von Umweltfaktoren wie Tiefe stimmten die eDNA- Daten bis zu 70 % mit den Fangdaten überein. Während eDNA-Methoden bisher vor allem für qualitative Erfassungen, also dem Nachweis von Arten, genutzt wurden, zeigt diese Studie deren Potenzial auch für semiquantitative Aussagen. Die Ergebnisse unterstreichen den Wert nicht-invasiver, kostengünstiger Methoden für ein nachhaltiges Fischereimanagement.
Die Ergebnisse der Studie sind publiziert:
Kasmi Y, Núñez-Riboni I, Blancke T, Möckel B, Bernreuther M, Stransky C, Hanel R (2025) Fish diversity assessment and semi-quantitative biomass estimation through metabarcoding of environmental DNA. Ecol Indic 173:113406, DOI:10.1016/j.ecolind.2025.113406